Le Washington Post via Getty Images
Josué Akey, université de Princeton
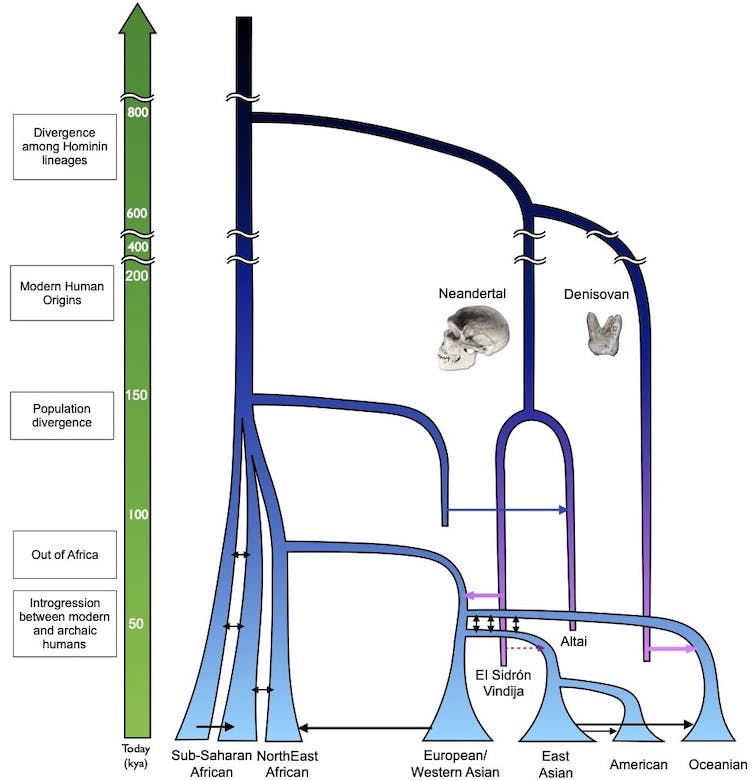
Lorsque les premiers humains modernes sont apparus en Afrique de l’Est il y a entre 200 000 et 300 000 ans, le monde était très différent de celui d’aujourd’hui. La plus grande différence était peut-être que nous – c’est-à-dire les gens de notre espèce, Homo sapiens – n’étaient que l’un des nombreux types d’humains (ou hominines) qui existaient simultanément sur Terre.
Des Néandertaliens bien connus et des Dénisoviens plus énigmatiques en Eurasie, au diminutif « hobbit » Homo floresiensis sur l’île de Flores en Indonésie, pour Homo naledi qui vivait en Afrique du Sud, de multiples homininés abondaient.
Puis, il y a entre 30 000 et 40 000 ans, tous les types de ces hominidés sauf un ont disparu, et pour la première fois nous étions seuls.
Jusqu’à récemment, l’un des mystères de l’histoire humaine était de savoir si nos ancêtres interagissaient et s’accouplaient avec ces autres types d’humains avant leur extinction. Cette question fascinante a fait l’objet de débats importants et souvent controversés entre scientifiques pendant des décennies, car les données nécessaires pour répondre à cette question n’existaient tout simplement pas. En fait, il semblait à beaucoup que les données n’existeraient jamais.
Svante Pääbo, cependant, prêtait peu d’attention à ce que les gens pensaient être ou n’était pas possible. Sa persévérance dans le développement d’outils pour extraire, séquencer et interpréter l’ADN ancien a permis de séquencer les génomes des Néandertaliens, des Denisoviens et des premiers humains modernes qui vivaient il y a plus de 45 000 ans.
Pour avoir développé ce nouveau domaine de la paléogénomique, Pääbo a reçu le prix Nobel 2022 de physiologie ou médecine. Cet honneur est non seulement une reconnaissance bien méritée pour les triomphes de Pääbo, mais aussi pour la génomique évolutive et les idées qu’elle peut contribuer à une compréhension plus complète de la santé et des maladies humaines.
Josué AkeyCC BY-ND
Mélange et accouplement, révélés par l’ADN
Les études génétiques des personnes vivantes au cours des dernières décennies ont révélé les contours généraux de l’histoire humaine. Notre espèce est née en Afrique, s’est dispersée à partir de ce continent il y a environ 60 000 ans, pour finalement se répandre dans presque tous les endroits habitables de la Terre. D’autres types d’humains existaient alors que les humains modernes migraient à travers le monde, mais les données génétiques montraient peu de preuves que les humains modernes s’accouplaient avec d’autres hominines.
Au cours de la dernière décennie, cependant, l’étude de l’ADN ancien, récupéré à partir de fossiles datant d’environ 400 000 ans, a révélé de nouveaux rebondissements surprenants dans l’histoire de l’humanité.
Par exemple, le génome de Néandertal a fourni les données nécessaires pour montrer définitivement que les humains et les Néandertaliens se sont accouplés. Les peuples non africains vivants aujourd’hui ont hérité d’environ 2% de leurs génomes d’ancêtres néandertaliens, grâce à ce type de métissage.
Dans l’une des plus grandes surprises, lorsque Pääbo et ses collègues ont séquencé l’ADN ancien obtenu à partir d’un petit fragment d’os de doigt supposé être néandertalien, il s’est avéré qu’il s’agissait d’un type d’humain totalement inconnu, maintenant appelé Denisovans. Les humains et les Dénisoviens se sont également accouplés, les niveaux les plus élevés d’ascendance dénisovienne étant présents aujourd’hui – entre 4% et 6% – chez les individus d’ascendance océanienne.
Étonnamment, l’ADN ancien d’une femme de 90 000 ans a révélé qu’elle avait une mère néandertalienne et un père denisovien. Bien qu’il reste encore de nombreuses questions sans réponse, l’image qui ressort des analyses de l’ADN ancien et moderne est que non seulement plusieurs hominidés se chevauchaient dans le temps et dans l’espace, mais que les accouplements étaient relativement courants.

alliance photo via Getty Images
Gènes archaïques que vous portez aujourd’hui
Estimer la proportion d’ascendance que les individus modernes ont des Néandertaliens ou des Dénisoviens est certainement intéressant. Mais les proportions d’ascendance fournissent des informations limitées sur les conséquences de ces accouplements anciens.
Par exemple, l’ADN hérité des Néandertaliens et des Denisoviens influence-t-il les fonctions biologiques qui se produisent dans nos cellules ? Cet ADN influence-t-il des traits comme la couleur des yeux ou la susceptibilité aux maladies ? Les séquences d’ADN de nos cousins évolutifs ont-elles déjà été bénéfiques, aidant les humains à s’adapter à de nouveaux environnements ?
Pour répondre à ces questions, nous devons identifier les fragments d’ADN néandertaliens et dénisoviens disséminés dans les génomes des individus modernes.
En 2014, mon groupe et le groupe de David Reich ont publié indépendamment les premières cartes des séquences néandertaliennes qui survivent dans l’ADN des humains modernes. Aujourd’hui, environ 40% du génome de Néandertal a été récupéré non pas en séquençant l’ADN ancien récupéré à partir d’un fossile, mais indirectement en reconstituant les séquences néandertaliennes qui persistent dans les génomes d’individus contemporains.
De même, en 2016, mon groupe et le groupe de David Reich ont publié les premiers catalogues complets de séquences d’ADN chez des individus modernes hérités d’ancêtres Denisovan. Étonnamment, lorsque nous avons analysé les séquences de Denisovan qui persistent chez les humains aujourd’hui, nous avons découvert qu’elles provenaient de deux populations Denisovan distinctes, et donc au moins deux vagues distinctes d’accouplements se sont produites entre les Denisovans et les humains modernes.
L’analyse de l’ADN de Néandertal et de Denisovan chez l’homme moderne révèle qu’une partie de leur séquence était nocive et a rapidement été purgée des génomes humains. En fait, la fraction initiale d’ascendance néandertalienne chez les humains qui vivaient il y a environ 45 000 ans était d’environ 10 %. Ce montant a rapidement diminué sur un petit nombre de générations pour atteindre les 2% observés chez les individus contemporains.
La suppression de séquences archaïques délétères a également créé de grandes régions du génome humain qui sont considérablement appauvries en ascendance néandertalienne et denisovienne. Ces déserts de séquences d’hominines archaïques sont intéressants car ils peuvent aider à identifier les changements génétiques qui contribuent à des traits humains modernes uniques, tels que notre capacité de langage, de pensée symbolique et de culture, bien qu’il y ait un débat sur le caractère unique de ces traits pour les humains modernes.
En revanche, il existe également des séquences héritées des Néandertaliens et des Dénisoviens qui étaient avantageuses et ont aidé les humains modernes à s’adapter à de nouveaux environnements alors qu’ils se dispersaient hors d’Afrique. Les versions néandertaliennes de plusieurs gènes liés au système immunitaire ont atteint une fréquence élevée dans plusieurs populations non africaines, ce qui a probablement aidé les humains à éviter l’exposition à de nouveaux agents pathogènes. De même, une version du APES1 Le gène, qui contribue à l’adaptation à haute altitude des populations tibétaines, a été hérité des Dénisoviens.
Il devient également clair que les séquences d’ADN héritées des ancêtres néandertaliens et dénisoviens contribuent au fardeau de la maladie chez les individus d’aujourd’hui. Il a été démontré que les séquences néandertaliennes influencent à la fois la sensibilité et la protection contre le COVID-19 sévère. Il a également été démontré que les séquences archaïques d’hominine influencent la susceptibilité à la dépression, au diabète de type 2 et à la maladie coeliaque, entre autres. Les études en cours en révéleront sans aucun doute davantage sur la contribution de l’ascendance néandertalienne et denisovienne aux maladies humaines.

Jens Schluete via Getty ImagesActualités
J’étais étudiant diplômé lorsque le Projet du génome humain touchait à sa fin il y a un peu plus de vingt ans. J’ai été attiré par la génétique parce que je trouvais fascinant qu’en analysant l’ADN d’individus d’aujourd’hui, on puisse découvrir des aspects de l’histoire d’une population qui s’est produite il y a des dizaines de milliers d’années.
Aujourd’hui, je suis tout aussi fasciné par les histoires contenues dans notre ADN, et le travail de Svante Pääbo et de ses collègues a permis de raconter ces histoires d’une manière qui n’était tout simplement pas possible auparavant.
Joshua Akey, professeur à l’Institut Lewis-Sigler de génomique intégrative, université de Princeton
Cet article est republié de The Conversation sous une licence Creative Commons. Lire l’article d’origine.

